cerna数据库,cerna机制
识别生物标志物是癌症研究cerna数据库的一个关键方向cerna数据库,今天小编就和大家分享一篇今年10月份发表在Frontiers in Oncology(IFcerna数据库:4.848)杂志,研究转移性结直肠癌相关ceRNA网络分析来识别生物标志物的文章。

基于lncRNA相关ceRNA网络分析识别结肠癌和直肠癌间致癌异质性的生物标志物
一.研究背景
结直肠癌(CRC)是全球癌症死亡的主要原因之一,此外,有新证据表明,结肠癌和直肠癌的风险因素和转移模式有很大差异。然而,这其中的致病差异的分子机制尚不清楚。因此,今天和大家介绍一篇基于lncRNA相关ceRNA网络(LceNET)识别非编码RNA生物标记物,来阐明结肠癌和直肠癌之间的致癌异质性的文章。
二.研究数据及 ***
1.数据cerna数据库:作者从TCGA-COAD和 TCGA-READ数据集下载459例结肠癌患者和171例直肠癌患者的RNA-Seq基因表达数据、miRNA-seq数据及临床数据。根据结直肠癌肿瘤节点转移(TNM)分期系统,将患者分为无远处转移(M0)和有远端转移(M1)两组。由于腺癌是CRC病例中最常见的组织型,占90%,因此仅保留具有RNA-seq和miRNA-seq数据的腺癌病例进行后续分析。
2.构建人类全局LceNETcerna数据库:作者首先从starBase、DIANA-Tarbase、miRTarbase及miRSponge数据库下载实验验证的 miRNA-mRNA关系对,接着作者从starBase、LncACTdb2.0、DIANALncBase Experimental及miRSponge数据库提取miRNA-lncRNA靶向关系。接着结合TCGA数据对这些靶向关系进行整合。此外,作者还通过超几何检验来评估每个lncRNA-mRNA对之间共有的miRNA的显著性,最终构建人类全局LceNET。
3.识别差异表达的mRNA、miRNA、lncRNA:为了与TCGA中的基因注释保持一致,作者首先利用GENCODE数据库对RNA-Seq表达数据中的lncRNAs和mRNA进行识别和注释。同时,miRNA-seq数据中miRNA参考miRBase数据库进行ID转换。然后,使用edgeR包装识别正常组织和M0/M1组织间差异表达的lncRNAs、mRNA和miRNAs。
4.对癌症的初始和转移重构背景特异的ceRNA网络:在ceRNA假设的基础上,作者采用两步构建背景特异的LceNETs。首先,将各组差异表达的lncRNA、miRNAs和mRNA分别映射到全局lncRNA相关的ceRNA网络中,提取背景相关的ceRNA网络。第二,使用匹配的表达谱,通过Pearson相关系数评估候选ceRNA三元组中lncRNA和mRNA的表达相关性。Hub节点定义为特定背景下LceNET中节点度前5%的节点。
5.差异表达mRNA的KEGG通路富集分析:作者使用ClusterProfiler包对差异表达mRNA进行KEGG功能富集分析。
6.生物信息学模型筛选lncRNA/miRNA生物标志物:研究应用一种新的生物信息学模型筛选结肠癌和直肠癌的候选lncRNA和miRNA生物标志物。在模型中,定义了NSM测度来检测lncRNA在特定背景ceRNA网络中竞争性结合miRNAs的能力。在起始或转移相关LceNETs中NSM值显著较高的lncRNA被认定为lncRNA的生物标志物。应用NSR在lncRNA-miRNA-mRNA三元网络中识别关键的miRNA。那些在起始或转移相关ceRNA网络中具有显著高NSR值的miRNA被识别为miRNA生物标志物。当lncRNA和miRNA被识别为生物标志物或者mRNA与肿瘤或者结直肠预后相关,则筛选其为关键的lncRNA-miRNA-mRNA三元组。
7.ROC分析:为了评估所识别的lncRNA和miRNA 生物标志物区分正常组织和癌症组织的敏感性和特异性,作者进行了ROC及AUC评估。
8.生存分析:为了评估lncRNA/miRNA生物标志物和关键mRNA的预后价值,作者将患者分为高表达组和低表达组,进行生存分析。
9.细胞系培养及qRT-PCR
三.研究的主要内容及结果
1.构建人类全局lncRNA相关ceRNA网络
在文章的第一部分,作者为了探索癌症中lncRNA相关的竞争性相互作用,使用计算 *** 来发现人类全基因组的ceRNA交互,并考虑了实验支持的miRNA-靶标对、ceRNA网络和TCGA数据库之间的lncRNA名称的一致性以及miRNA调控相似性等因素。最终在识别出的ceRNA互作三元组的基础上,构建了由3102个lncRNA、1085个miRNAs和16490个mRNA组成的全局LceNET,研究的流程如图1所示。
图1 ceRNA网络构建及刻画流程
2.结肠癌和直肠癌中差异表达RNA的研究
在这一部分,作者计算了结肠癌和直肠癌远端转移及未远端转移组与对照组的差异表达miRNA、mRNA及lncRNA。在这四组中,共有271个lncRNAs、198个miRNAs和980个mRNA被识别为共出现的DeRNAs(图2A)。接着作者对差异RNA进行功能富集复分析,结果如2B-E所示。
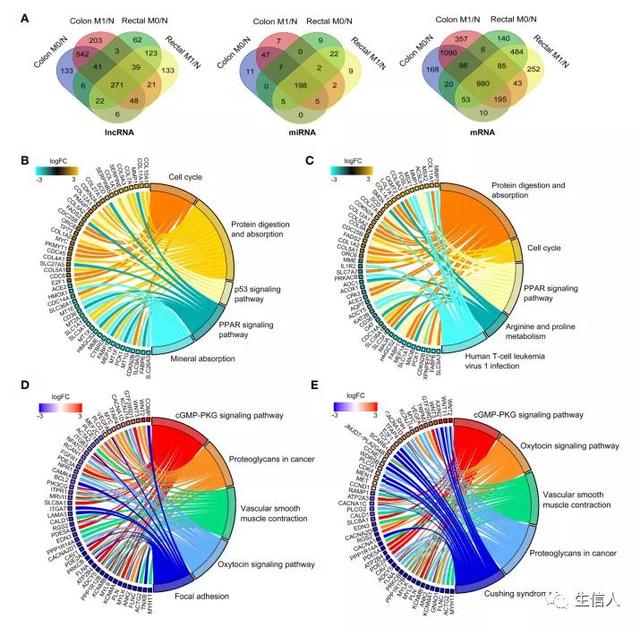
图2 结肠癌和直肠癌中差异表达RNA的全局概况
3.在结肠癌和直肠癌的发生和转移过程中lncRNA和mRNAs之间的动态竞争相互作用
在这一部分作者通过将结肠M0/N、结肠M1/N、直肠M0/N和直肠M1/N组的DeRNAs映射到全局人类LceNETs中,提取lncRNA-miRNA-mRNA相互作用,从而建立背景特异的LceNETs。作者构建的四个背景特异LceNETs如图3A所示,可以观察到每个LceNET中的大部分ceRNA节点都是相互连接的,并且可以通过共享的miRNAs相互交叉通信,说明调控关系是复杂而稳健的。因此,关键的ceRNA相互作用的扰动可能会影响整个LceNET的稳定性。接下来通过拓扑分析观察到四个网络的节点度分布遵循幂律分布,表明LceNETs是无标度网络,而不是随机连接的网络。此外,作者对四个网络中的hub节点进行分析,这些节点可能在调节结直肠癌发生方面具有重要的潜力。
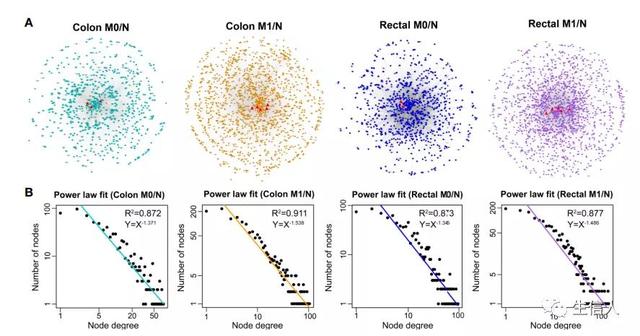
图3 背景特异的ceRNA网络及度分布
4.LceNET驱动的lncRNA生物标志物在结肠癌和直肠癌的发生中的诊断和预后意义
在这一部分,作者开发了一种计算 *** ,通过共享miRNAs (NSM)的数量来评估lncRNA作为ceRNA的潜力,并基于背景特异的LceNETs来探索参与结肠癌和直肠癌启动和转移的lncRNA生物标志物。如图4A所示,与对应背景特异LceNETs中的所有lncRNA相比,生物标志物lncRNA 的NSM值明显更高。此外,通过ROC曲线分析,评估生物标志物lncRNA在诊断无转移或有远处转移的癌症患者中的敏感性和特异性,结果如图4B,C所示,可以看出生物标志物lncRNA具有很好的效能。其中,lncRNA ZFAS1在结肠M1/N组和直肠M1/N组中被识别为特异性的生物标志物,表明其可用于预测CRC患者的远端转移(表1)。接着作者按生物标志物lncRNA表达的高低进行生存分析,发现MALAT1和KCNQ1OT1与结肠癌患者的总生存(OS)时间显著相关(图4D)。由于lncRNAs KCNQ1OT1和SNHG1在诊断结肠癌或直肠癌患者方面表现出很高的潜力,作者通过已发表的GEO数据集GSE21510、GSE23878和GSE9348进一步验证了其实验基因表达模式和诊断性能。结果发现KCNQ1OT1和SNHG1在肿瘤组织中均显著上调(图5A、B),并具有优异的结肠直肠癌患者识别能力(图5C)。且KCNQ1OT1和SNHG1在CRC细胞系中的表达水平明显高于正常结肠上皮细胞系NCM460(图5E)。此外,CCLE数据库和qRT-PCR分析结果也显示,KCNQ1OT1和SNHG1在高转移潜能CRC细胞系中的表达水平明显高于低转移潜能CRC细胞系(图5D,E),表明了其促进CRC转移的作用。
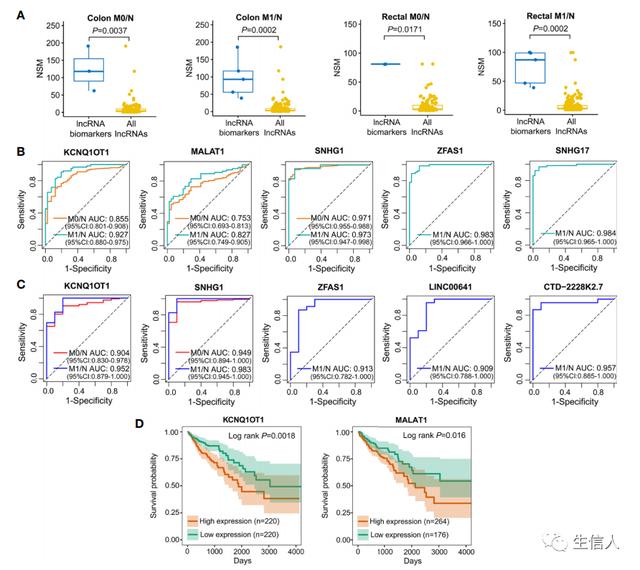
图4 lncRNA生物标志物在结肠癌和直肠癌的发生和转移中的作用
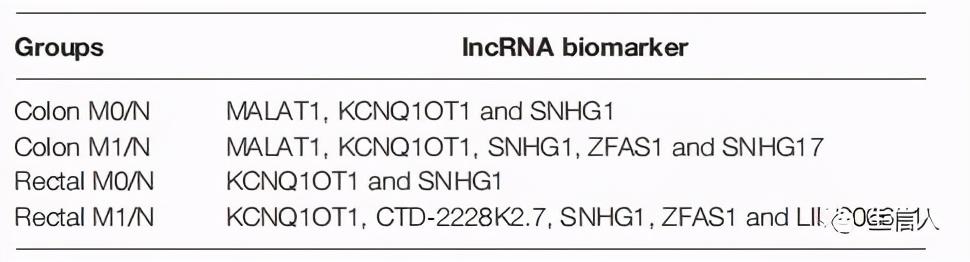
表1 lncRNA生物标志物参与结肠直肠癌的癌变
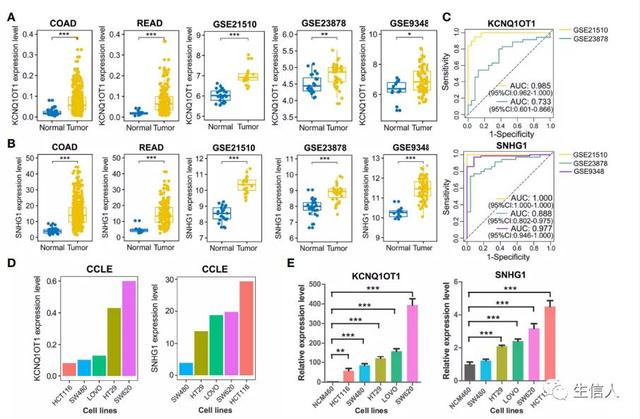
图5 通过评估lncRNA的表达模式和诊断性能来验证常见的lncRNA生物标志物
5.在LceNETs中识别的表明结肠癌和直肠癌的致癌的miRNA生物标记物的特征
由于越来越多的证据表明,多个miRNAs可以作为生物标志物来指导癌症诊断和治疗的决策,因此在这一部分作者采用 single-regulatory理论将单线调控的miRNA -靶点相互作用视为生物网络的脆弱结构,在ceRNA网络中识别miRNA生物标志物。如图6A所示,可以观察到与背景特异LceNETs中的所有miRNA相比,miRNA生物标志物具有显著更高的NSR值。在图6B中可以观察到肿瘤和正常组织之间这些生物标志物miRNA的差异表达,接着作者对这些生物标志物miRNA进行ROC,AUC评估(图6C,D),发现它们能很好地区分正常和无转移或远端转移的肿瘤。
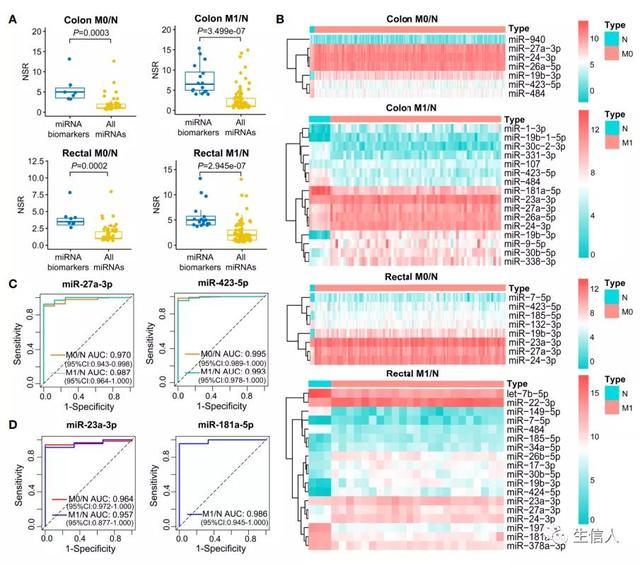
图6 miRNA在结肠癌和直肠癌的发生和转移中的特征研究
6.关键的lncRNA-miRNA-mRNA互作在结肠和直肠癌中的致癌作用
在文章的最后一部分,作者为了进一步阐明结肠癌和直肠癌的分子差异,通过筛选lncRNA和miRNA生物标志物组成的相互作用,识别了关键的参与肿瘤起始和转移的与lncRNA相关的竞争性三元组。由于miRNA负调控靶基因表达,只有miRNA及其lncRNA/mRNA靶基因表达模式相反的三元组被保留(图7A)。结果发现识别的ceRNA三元组中ANKRD36、FKBP14、PPRC1及NAB1与结肠癌患者的预后密切相关(图7B),而PCGF2和ATP6V1F与直肠癌患者的预后紧密相连(图7C),表明lncRNA生物标志物可能通过ceRNA机制调控预后相关基因导致CRC发生和转移。此外,作者还对所识别的ceRNA三元组中lncRNA与代表性靶mRNA的相关性进行了研究,结果发现,在结肠M0/N组、结肠M1/N组、直肠M0/N组和直肠M1/N组中,KCNQ1OT1与ANKRD36、FKBP14、OGT和PCGF2呈强相关,SNHG1与ORC6、ORC6、EZH2和ATP6V1F呈强相关(图8A)。接着通过分析显著富集的KEGG通路,发现KCNQ1OT1调控的COL12A1和PCGF2分别与蛋白质消化吸收和轴突引导通路密切相关。相比之下,ORC6和ATP6V1F分别在细胞周期和过氧化物酶体通路中富集。基于以上结果,作者提出了KCNQ1OT1和SNHG1作为ceRNA在结肠癌和直肠癌中可能的调控机制(图8B, C)。
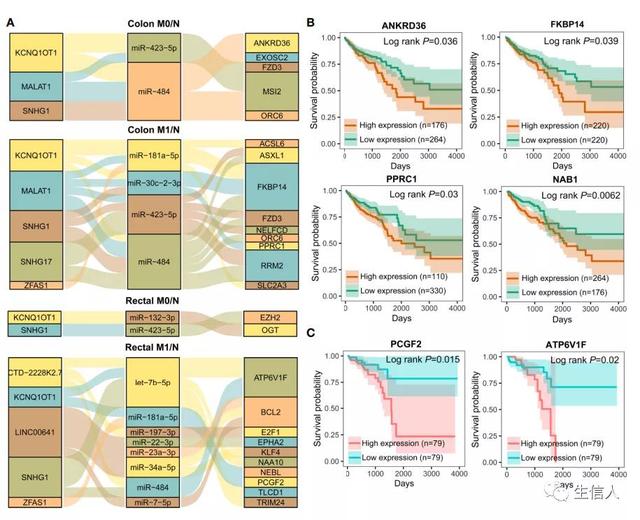
图7 lncRNA-miRNA生物标志物组成的lncRNA-miRNA- mRNA互作及靶mRNA的预后价值
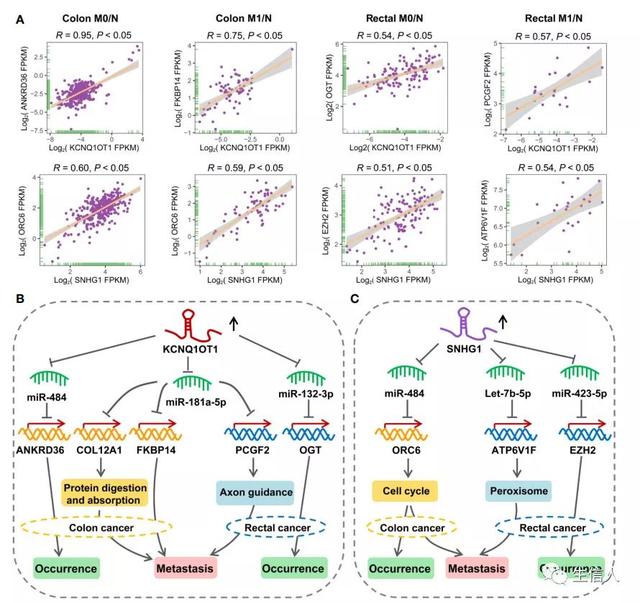
图8 KCNQ1OT1和SNHG1在结肠癌和直肠癌发生和转移中的调控机制
到这里,这篇文章的主要内容就介绍完了,文章通过整合多个数据库的数据资源构建了结直肠癌的ceRNA网络,并使用差异分析、功能富集、生存分析等生物信息 *** 对网络中的节点进行了分析。同时结合其他数据资源及 *** 对结果进行了验证。文章从多个角度对ceRNA网络进行了刻画,使基本的ceRNA研究内容变得充实,研究的角度及 *** 值得参考学习。